2.1 데이터 전처리 기초#
이 절에서는 데이터를 본격적으로 분석하기 이전에 다음과 같은 패키지를 사용하여 기초적인 전처리(preprocessing)를 하는 방법을 설명한다.
missingno 패키지: 결측 데이터 검색
sklearn.impute 패키지: 결측 데이터 대체
patsy 패키지: 데이터 선택, 변환, 추가, 스케일링
sklearn.preprocessing 패키지: 스케일링, 변환
missingno 패키지#
현실에서 데이터를 수집하다보면 데이터의 일부를 얻지 못하거나 누락되는 결측(missing) 데이터가 생긴다. missingno 패키지는 pandas 데이터프레임에서 결측 데이터를 찾는 기능을 제공한다. 이 패키지를 사용하려면 데이터프레임에 결측 데이터가 NaN(not a number) 값으로 저장되어 있어야 한다.
주의할 점은 NaN값은 부동소수점 실수 자료형에만 있는 값이므로 정수 자료를 데이터프레임에 넣을 때는 Int64Dtype 자료형을 명시해주어야 하고 시간 자료형을 넣을 때도 parse_dates 인수로 날짜시간형 파싱을 해주어야 datetime64[ns] 자료형이 되어 결측 데이터가 NaT(not a time) 값으로 표시된다.
from io import StringIO
csv_data = StringIO("""
x1,x2,x3,x4,x5
1,0.1,"1",2019-01-01,A
2,,,2019-01-02,B
3,,"3",2019-01-03,C
,0.4,"4",2019-01-04,A
5,0.5,"5",2019-01-05,B
,,,2019-01-06,C
7,0.7,"7",,A
8,0.8,"8",2019-01-08,B
9,0.9,,2019-01-09,C
""")
df = pd.read_csv(csv_data, dtype={"x1": pd.Int64Dtype()}, parse_dates=[3])
df
| x1 | x2 | x3 | x4 | x5 | |
|---|---|---|---|---|---|
| 0 | 1 | 0.1 | 1.0 | 2019-01-01 | A |
| 1 | 2 | NaN | NaN | 2019-01-02 | B |
| 2 | 3 | NaN | 3.0 | 2019-01-03 | C |
| 3 | NaN | 0.4 | 4.0 | 2019-01-04 | A |
| 4 | 5 | 0.5 | 5.0 | 2019-01-05 | B |
| 5 | NaN | NaN | NaN | 2019-01-06 | C |
| 6 | 7 | 0.7 | 7.0 | NaT | A |
| 7 | 8 | 0.8 | 8.0 | 2019-01-08 | B |
| 8 | 9 | 0.9 | NaN | 2019-01-09 | C |
판다스 데이터프레임 자체도 isnull() 또는 isna() 메서드로 사용하면 결측 데이터의 위치를 알아내는 것이 가능하다.
df.isnull()
| x1 | x2 | x3 | x4 | x5 | |
|---|---|---|---|---|---|
| 0 | False | False | False | False | False |
| 1 | False | True | True | False | False |
| 2 | False | True | False | False | False |
| 3 | True | False | False | False | False |
| 4 | False | False | False | False | False |
| 5 | True | True | True | False | False |
| 6 | False | False | False | True | False |
| 7 | False | False | False | False | False |
| 8 | False | False | True | False | False |
하지만 데이터가 많은 경우에는 일일히 확인할 수 없으므로 기껏해야 sum() 메서드를 결합하여 결측 데이터의 갯수를 유추하는 것만 가능하다.
df.isnull().sum()
x1 2
x2 3
x3 3
x4 1
x5 0
dtype: int64
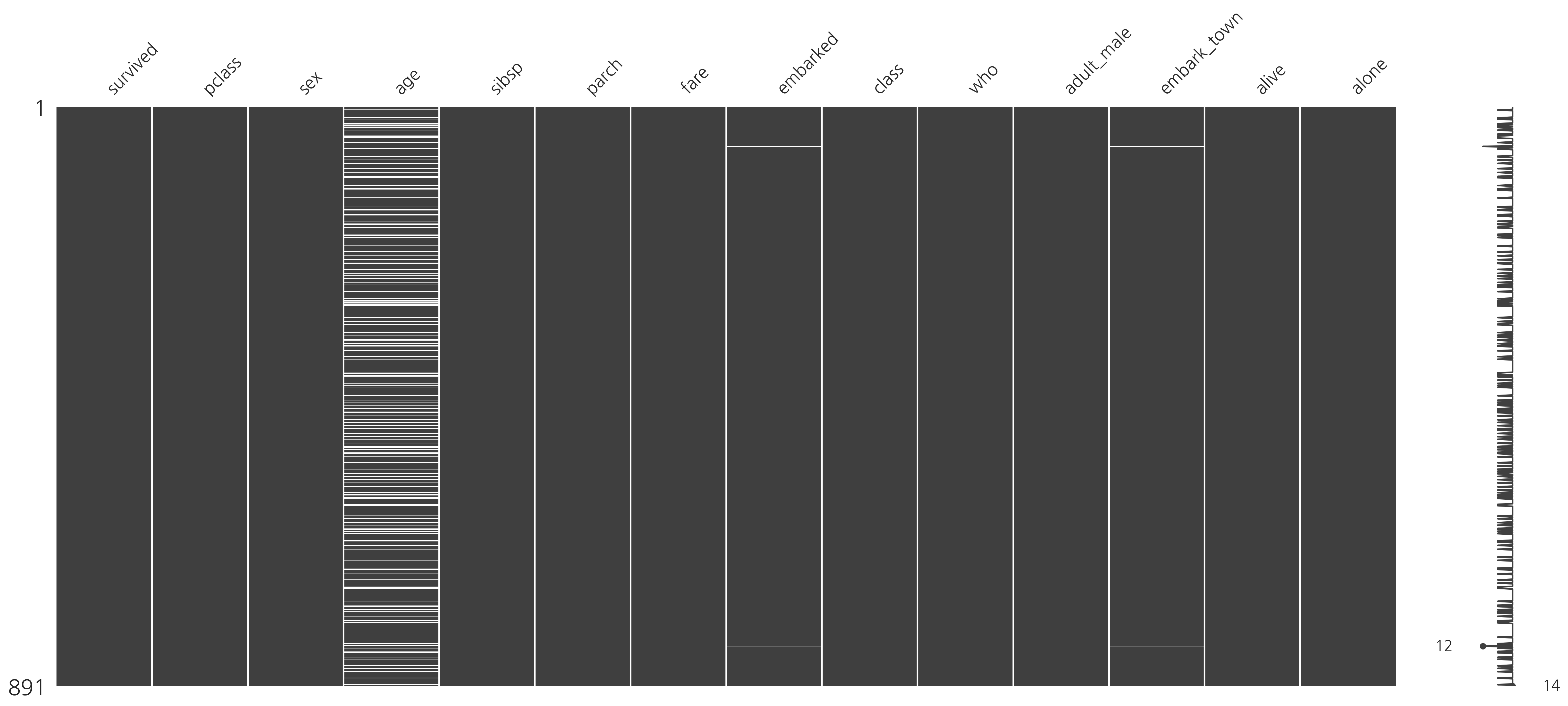
하지만 missingno 패키지를 사용하면 대규모의 데이터에서도 결측 데이터에 대한 시각화를 쉽게 할 수 있다. matrix() 함수는 결측 데이터를 시각화하는 명령이다. 결측된 데이터는 흰색으로, 그렇지 않은 데이터는 검은색으로 나타난다. 가장 오른쪽에 있는 것은 스파크라인(spark line)이라고 부르고, 각 행의 데이터 완성도를 표현한다.
import missingno as msno
msno.matrix(df)
plt.show()

만약 각 열에 결측 데이터가 얼마나 존재하는지 시각화 하고 싶다면, bar() 함수를 사용한다.
msno.bar(df)
plt.show()
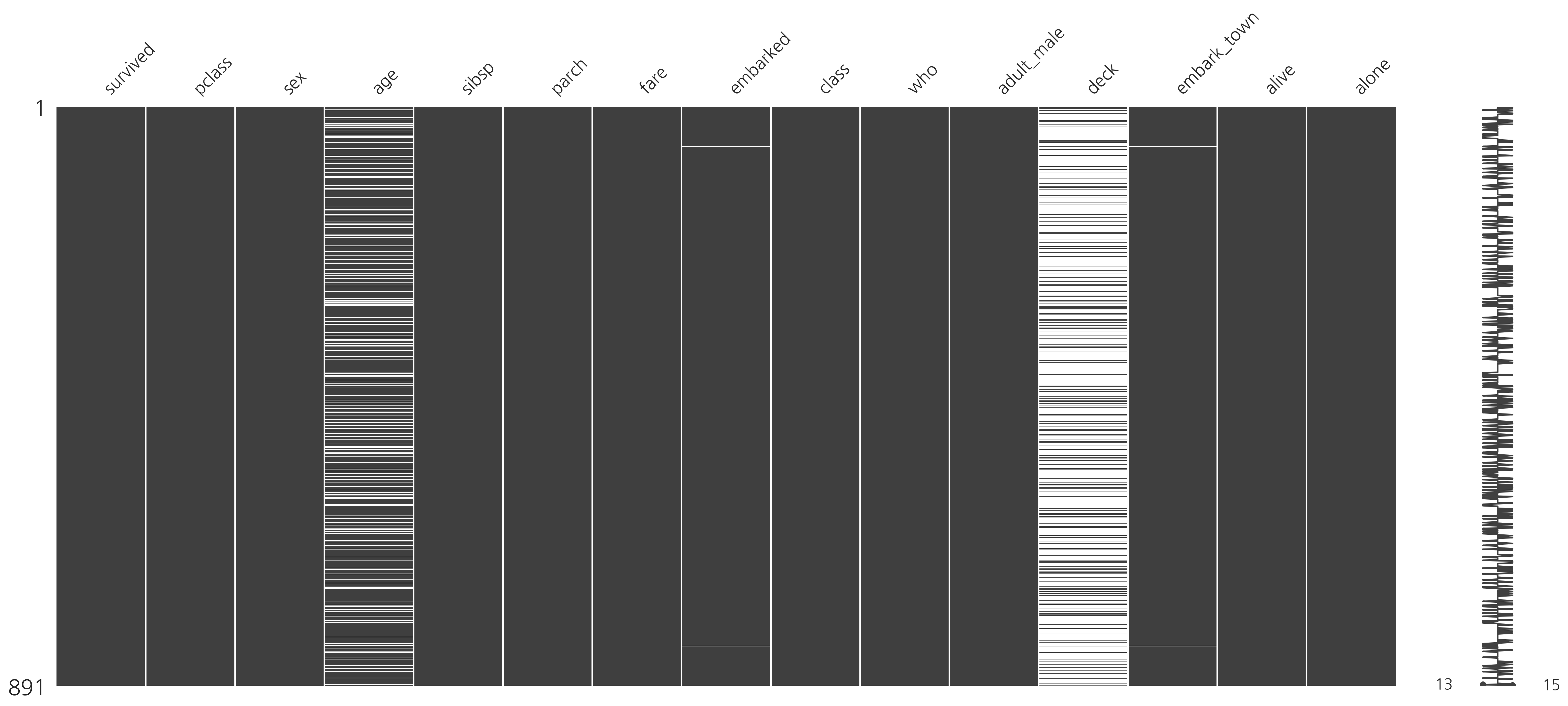
대규모 데이터에서 missingno 패키지가 어떻게 쓰일 수 있는지 살펴보기 위해 타이나닉 생존자 데이터를 예로 들어본다. 타이타닉 생존자 데이터는 seaborn 패키지에서 제공하는 예제 데이터다.
titanic = sns.load_dataset("titanic")
titanic.tail()
| survived | pclass | sex | age | sibsp | parch | fare | embarked | class | who | adult_male | deck | embark_town | alive | alone | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 886 | 0 | 2 | male | 27.0 | 0 | 0 | 13.00 | S | Second | man | True | NaN | Southampton | no | True |
| 887 | 1 | 1 | female | 19.0 | 0 | 0 | 30.00 | S | First | woman | False | B | Southampton | yes | True |
| 888 | 0 | 3 | female | NaN | 1 | 2 | 23.45 | S | Third | woman | False | NaN | Southampton | no | False |
| 889 | 1 | 1 | male | 26.0 | 0 | 0 | 30.00 | C | First | man | True | C | Cherbourg | yes | True |
| 890 | 0 | 3 | male | 32.0 | 0 | 0 | 7.75 | Q | Third | man | True | NaN | Queenstown | no | True |
각 열이 의미하는 바는 다음과 같다.
survived : 생존 여부
pclass : 승객의 클래스
sex : 성별. male, female로 표기
sibsp : 형제 혹은 자매의 수
parch : 부모 혹은 자녀의 수
fare : 탑승 요금
embarked : 출발지의 고유 이니셜
class : 선실의 클래스
who : male, female을 man, woman으로 표기
adult_male : 성인 남성 인지 아닌지 여부
deck : 선실 고유 번호의 가장 앞자리 알파벳(A ~ G)
embark_town : 출발지
alive : 생존 여부 데이터를 yes 혹은 no로 표기
alone : 가족이 없는 경우 True
missingno를 이용하여 타이타닉 데이터를 살펴보면 age, deck, embarkd, embark_town 열 등에 결측 데이터가 있는 것을 볼 수 있다.
msno.matrix(titanic)
plt.show()
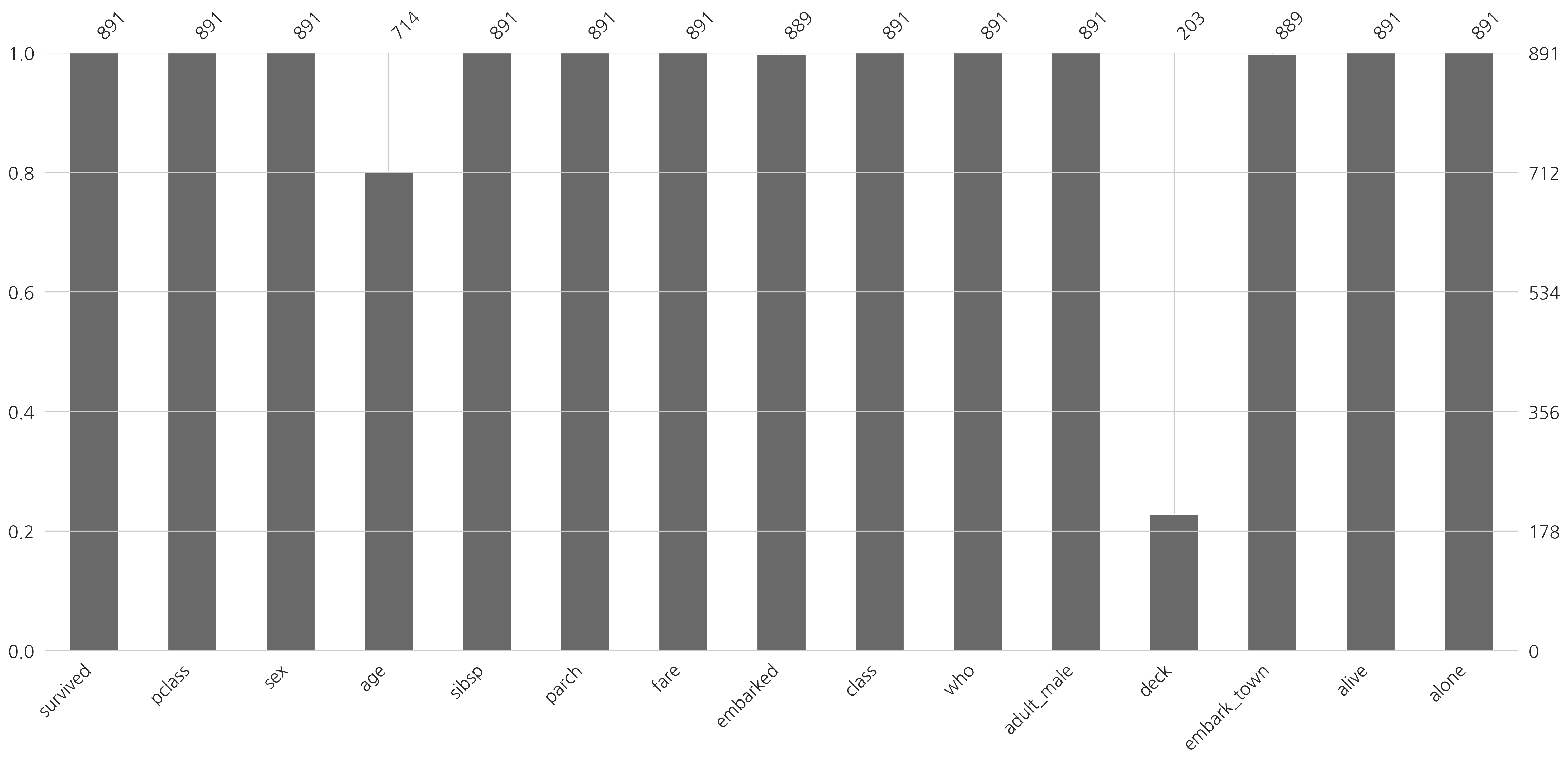
msno.bar(titanic)
plt.show()
결측된 데이터를 처리하는 방법은 두 가지다.
결측된 데이터가 너무 많은 경우 해당 데이터 열 전체를 삭제할 수 있다.
결측된 데이터가 일부인 경우 가장 그럴듯한 값으로 대체할 수 있다. 이를 **결측 데이터 대체(imputation)**라고 한다.
판다스의 dropna() 메서드를 사용하면 결측 데이터가 존재하는 행이나 열을 지울 수 있다. 다음 코드는 위에서 예로 든 df 데이터프레임에서 결측 데이터가 존재하는 모든 행을 지운다.
df.dropna()
| x1 | x2 | x3 | x4 | x5 | |
|---|---|---|---|---|---|
| 0 | 1 | 0.1 | 1.0 | 2019-01-01 | A |
| 4 | 5 | 0.5 | 5.0 | 2019-01-05 | B |
| 7 | 8 | 0.8 | 8.0 | 2019-01-08 | B |
axis 인수를 1로 설정하면 결측 데이터가 있는 열을 제거한다.
df.dropna(axis=1)
| x5 | |
|---|---|
| 0 | A |
| 1 | B |
| 2 | C |
| 3 | A |
| 4 | B |
| 5 | C |
| 6 | A |
| 7 | B |
| 8 | C |
thresh 인수를 사용하면 특정 갯수 이상의 비결측 데이터가 있는 행 또는 열만 남긴다.
df.dropna(thresh=7, axis=1)
| x1 | x4 | x5 | |
|---|---|---|---|
| 0 | 1 | 2019-01-01 | A |
| 1 | 2 | 2019-01-02 | B |
| 2 | 3 | 2019-01-03 | C |
| 3 | NaN | 2019-01-04 | A |
| 4 | 5 | 2019-01-05 | B |
| 5 | NaN | 2019-01-06 | C |
| 6 | 7 | NaT | A |
| 7 | 8 | 2019-01-08 | B |
| 8 | 9 | 2019-01-09 | C |
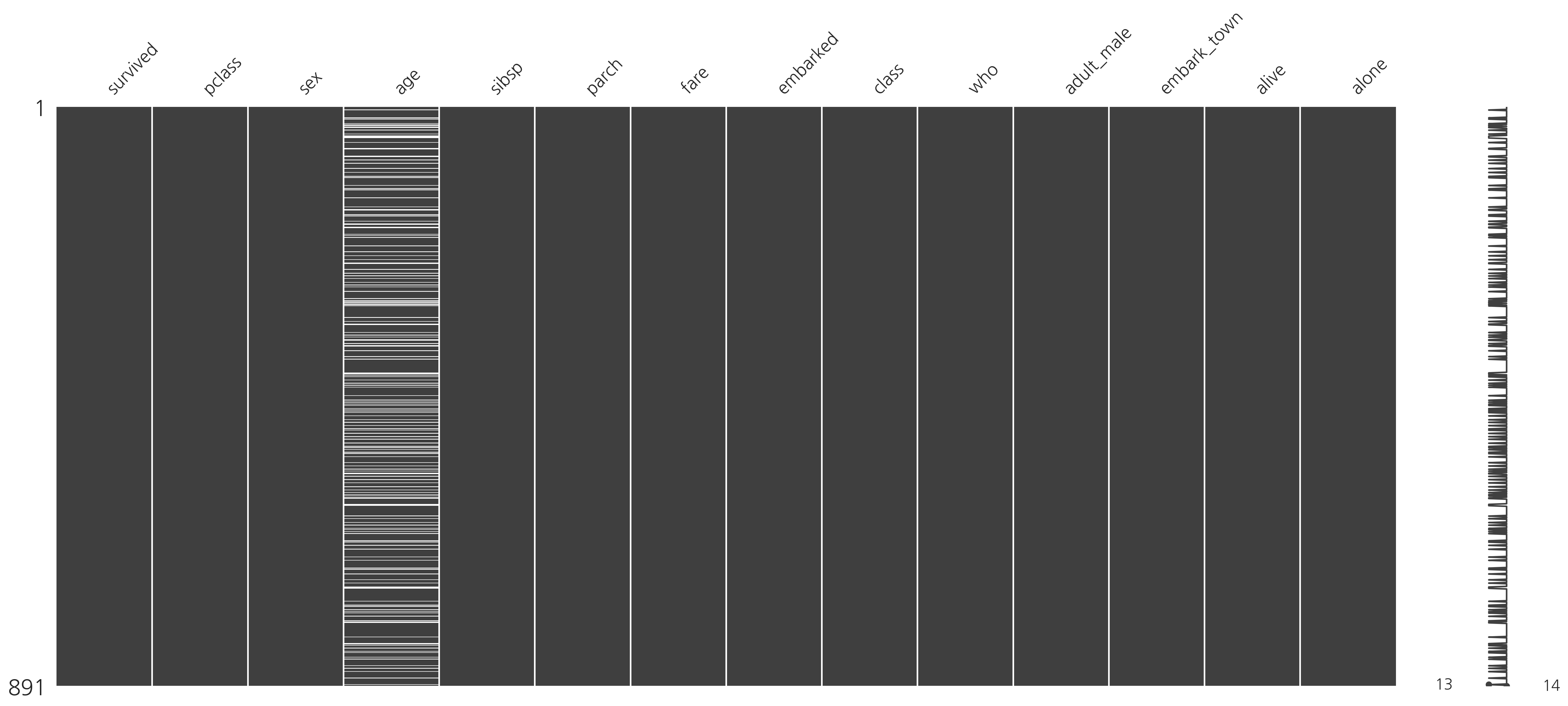
타이타닉 데이터에서 deck 데이터는 결측된 데이터가 너무 많기 때문에 이 방법으로 데이터를 삭제한다.
# 데이터가 절반 이상이 없는 열을 삭제
titanic = titanic.dropna(thresh=int(len(titanic) * 0.5), axis=1)
msno.matrix(titanic)
plt.show()
결측 데이터를 대체할 때는 해당 열의 비결측 데이터의 평균값 혹은 중앙값 등을 대체값으로 사용하여 결측된 데이터를 채운다. scikit-learn 패키지의 SimpleImputer 클래스를 사용하면 쉽게 결측 데이터를 대체할 수 있다. 사용법은 다음과 같다.
SimpleImputer클래스 객체를 생성한다. 이 대strategy인수를"mean"으로 하면 평균값"median"으로 하면 중앙값,"most_frequent"로 하면 최빈값을 대체값으로 사용한다.fit_transform메서드를 사용하여 대체값이 채워신 데이터프레임을 생성한다.
strategy 인수를 선택하는 방법은 다음과 같다.
데이터가 실수 연속값인 경우에는 평균 또는 중앙값을 사용할 수 있다. 값의 분포가 대칭적이면 평균이 좋고 값의 분포가 심하게 비대칭인 경우에는 중앙값이 적당하다.
데이터가 범주값이거나 정수값인 경우에는 최빈값을 사용할 수 있다.
from sklearn.impute import SimpleImputer
imputer = SimpleImputer(strategy="most_frequent")
df = pd.DataFrame(imputer.fit_transform(df), columns=df.columns)
df
| x1 | x2 | x3 | x4 | x5 | |
|---|---|---|---|---|---|
| 0 | 1 | 0.1 | 1 | 2019-01-01 | A |
| 1 | 2 | 0.1 | 1 | 2019-01-02 | B |
| 2 | 3 | 0.1 | 3 | 2019-01-03 | C |
| 3 | 1 | 0.4 | 4 | 2019-01-04 | A |
| 4 | 5 | 0.5 | 5 | 2019-01-05 | B |
| 5 | 1 | 0.1 | 1 | 2019-01-06 | C |
| 6 | 7 | 0.7 | 7 | 2019-01-01 | A |
| 7 | 8 | 0.8 | 8 | 2019-01-08 | B |
| 8 | 9 | 0.9 | 1 | 2019-01-09 | C |
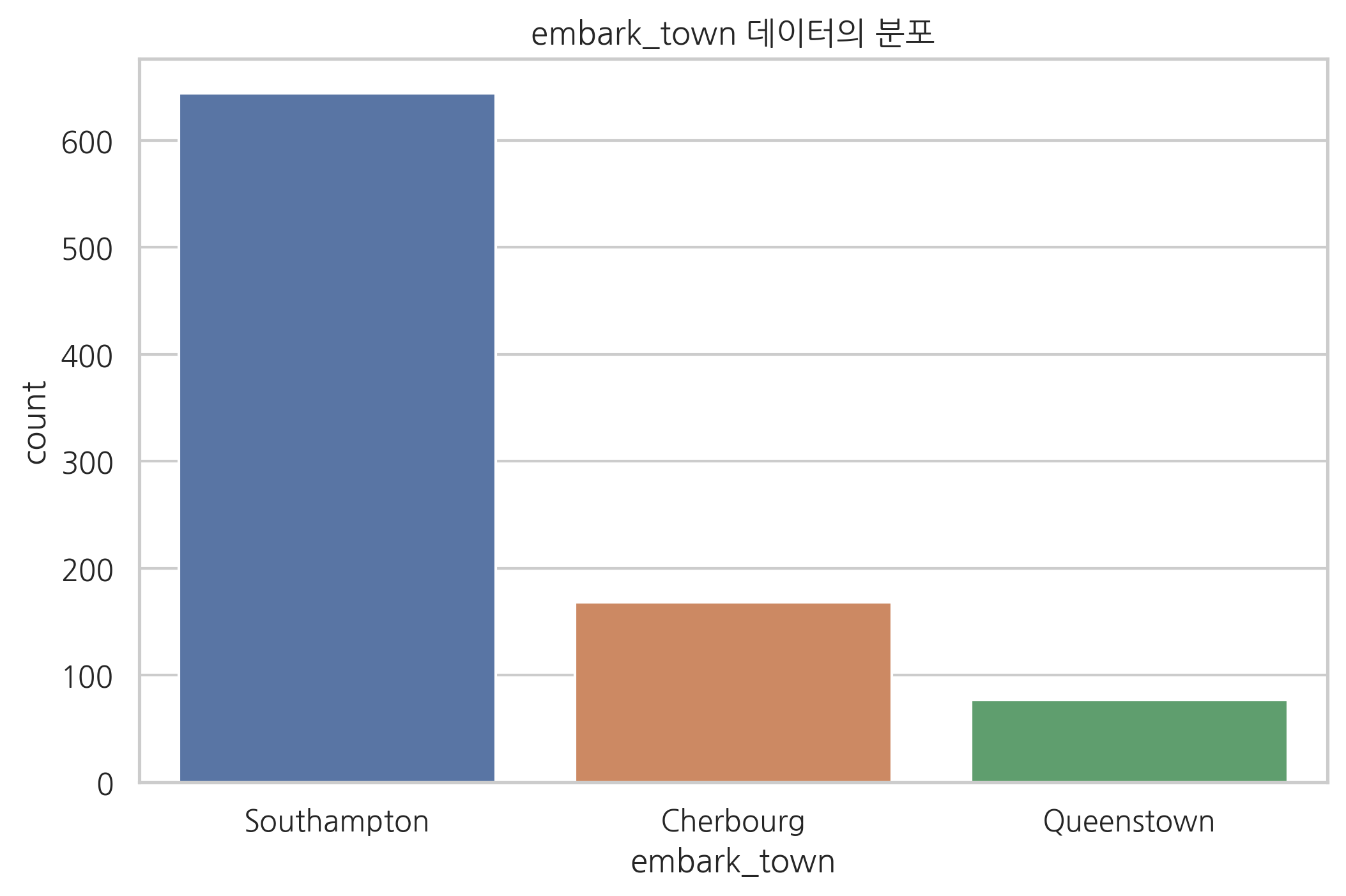
이번에는 타이타닉 데이터에서 embark_town 데이터부터 대체해보자. 이 값은 범주값이므로 strategy="most_frequent"로 하여 최빈값을 대체값으로 사용한다. 대체하기 전의 embark_town 데이터 분포를 살펴보면 Southhamton 값이 가장 많다. 대체 후에는 이 값으로 결측 데이터가 대체될 것이다.
sns.countplot(titanic.embark_town)
plt.title("embark_town 데이터의 분포")
plt.show()
SimpleImputer 클래스로 대체를 한 결과는 다음과 같다. embarked 데이터도 사실상 같은 데이터이므로 같이 대체한다.
from sklearn.impute import SimpleImputer
imputer_embark_town = SimpleImputer(strategy="most_frequent")
titanic["embark_town"] = imputer_embark_town.fit_transform(titanic[["embark_town"]])
titanic["embarked"] = imputer_embark_town.fit_transform(titanic[["embarked"]])
msno.matrix(titanic)
plt.show()
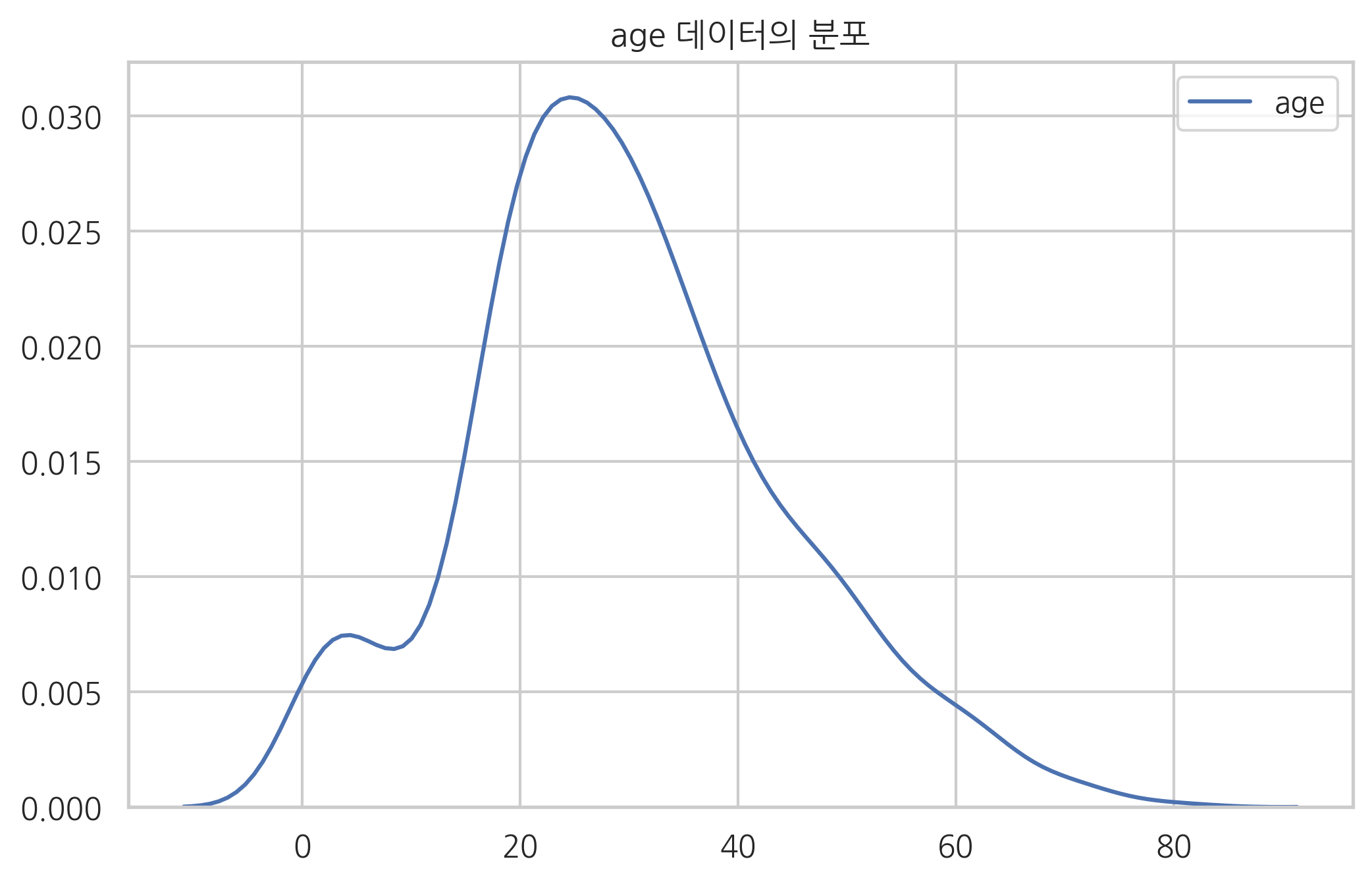
다음으로 age 데이터를 대체한다. 대체하기 전의 age 데이터 분포를 살펴보면 비대칭인 것을 볼 수 있다.
sns.kdeplot(titanic.age)
plt.title("age 데이터의 분포")
plt.show()
따라서 strategy="median"로 하여 중앙값을 대체값으로 사용한다. 다음 그림에서 결측값이 사라진 것을 확인할 수 있다.
imputer_age = SimpleImputer(strategy="median")
titanic["age"] = imputer_embark_town.fit_transform(titanic[["age"]])
msno.matrix(titanic)
plt.show()
patsy 패키지#
이번에는 patsy 패키지를 사용하여 데이터프레임에서 원하는 데이터만 선택하거나 새로운 데이터를 조합 생성하는 방법을 살펴본다. 설명을 위해 patsy 패키지가 제공하는 demo_data() 함수로 다음과 같은 예제 데이터프레임을 만들자. demo_data() 함수는 x로 시작하는 변수에 대해 임의의 실수 데이터를 생성한다.
from patsy import demo_data
df = pd.DataFrame(demo_data("x1", "x2", "x3", "x4", "x5"))
df
| x1 | x2 | x3 | x4 | x5 | |
|---|---|---|---|---|---|
| 0 | 1.764052 | -0.977278 | 0.144044 | 0.333674 | -2.552990 |
| 1 | 0.400157 | 0.950088 | 1.454274 | 1.494079 | 0.653619 |
| 2 | 0.978738 | -0.151357 | 0.761038 | -0.205158 | 0.864436 |
| 3 | 2.240893 | -0.103219 | 0.121675 | 0.313068 | -0.742165 |
| 4 | 1.867558 | 0.410599 | 0.443863 | -0.854096 | 2.269755 |
데이터 분석을 시작할 때는 다양한 데이터를 하나의 데이터프레임에 넣고 시작하는 경우가 많다. patsy 패키지가 제공하는 dmatrx() 함수를 사용하면 데이터 프레임에 상수항을 추가하거나 원하는 데이터만 선택하거나 변형할 수 있다. dmatrx() 함수에 모형 정의 문자열 formula와 원 데이터를 담은 데이터프레임 data을 입력하면 formula에서 지정한 대로 변환된 데이터 data_transformed를 출력한다.
data_transformed = dmatrix(formula, data)
formula는 데이터 열 이름 기반으로 구성된 문자열이다. 선택하고자 하는 데이터 열 이름을 +로 연결한 formula 문자열을 입려하면 자동으로 해당 데이터만 뽑아준다. 예를 들어 전체 데이터 중 x1만을 뽑고 싶으면 formula 문자열에 x1 + 0이라고 입력한다.
from patsy import dmatrix
dmatrix("x1 + 0", data=df)
DesignMatrix with shape (5, 1)
x1
1.76405
0.40016
0.97874
2.24089
1.86756
Terms:
'x1' (column 0)
전체 데이터 중 x1, x2, x3를 뽑고 싶으면 formula 문자열에 x1 + x2 + x3 + 0이라고 입력한다.
dmatrix("x1 + x2 + x3 + 0", data=df)
DesignMatrix with shape (5, 3)
x1 x2 x3
1.76405 -0.97728 0.14404
0.40016 0.95009 1.45427
0.97874 -0.15136 0.76104
2.24089 -0.10322 0.12168
1.86756 0.41060 0.44386
Terms:
'x1' (column 0)
'x2' (column 1)
'x3' (column 2)
+ 0은 1로 구성된 상수항을 넣지 말라는 뜻이다. 만약 이 부분이 없으면 patsy는 자동으로 가장 앞에 Intercept란 이름의 상수항 데이터를 추가한다.
dmatrix("x1 + x2 + x3", data=df)
DesignMatrix with shape (5, 4)
Intercept x1 x2 x3
1 1.76405 -0.97728 0.14404
1 0.40016 0.95009 1.45427
1 0.97874 -0.15136 0.76104
1 2.24089 -0.10322 0.12168
1 1.86756 0.41060 0.44386
Terms:
'Intercept' (column 0)
'x1' (column 1)
'x2' (column 2)
'x3' (column 3)
- 1도 + 0과 같은 뜻이다.
dmatrix("x1 + x2 + x3 - 1", data=df)
DesignMatrix with shape (5, 3)
x1 x2 x3
1.76405 -0.97728 0.14404
0.40016 0.95009 1.45427
0.97874 -0.15136 0.76104
2.24089 -0.10322 0.12168
1.86756 0.41060 0.44386
Terms:
'x1' (column 0)
'x2' (column 1)
'x3' (column 2)
dmatrix() 함수는 변수를 어떤 함수에 넣어서 다른 갑으로 만드는 수학 변환(transform)도 가능하다.
dmatrix("x1 + np.log(np.abs(x2))", df)
DesignMatrix with shape (5, 3)
Intercept x1 np.log(np.abs(x2))
1 1.76405 -0.02298
1 0.40016 -0.05120
1 0.97874 -1.88811
1 2.24089 -2.27090
1 1.86756 -0.89014
Terms:
'Intercept' (column 0)
'x1' (column 1)
'np.log(np.abs(x2))' (column 2)
numpy 함수 뿐 아니라 사용자 정의 함수도 사용할 수 있다.
def ten_times(x):
return 10 * x
dmatrix("ten_times(x1)", df)
DesignMatrix with shape (5, 2)
Intercept ten_times(x1)
1 17.64052
1 4.00157
1 9.78738
1 22.40893
1 18.67558
Terms:
'Intercept' (column 0)
'ten_times(x1)' (column 1)
기존의 데이터를 조합 연산하여 새로운 데이터를 만드는 것도 가능하다. 특히 두 변수를 곱해서 만들어지는 새로운 변수를 **상호작용(interaction)**이라고 한다. 상호작용은 : 기호를 사용하여 만들어진다.
dmatrix("x1 + x2 + x1:x2 + 0", df)
DesignMatrix with shape (5, 3)
x1 x2 x1:x2
1.76405 -0.97728 -1.72397
0.40016 0.95009 0.38018
0.97874 -0.15136 -0.14814
2.24089 -0.10322 -0.23130
1.86756 0.41060 0.76682
Terms:
'x1' (column 0)
'x2' (column 1)
'x1:x2' (column 2)
두 변수와 상호작용을 한꺼번에 표시할 때는 *를 사용한다. 따라서 다음 수식은 바로 전의 수식과 동일하다.
dmatrix("x1 * x2 + 0", df)
DesignMatrix with shape (5, 3)
x1 x2 x1:x2
1.76405 -0.97728 -1.72397
0.40016 0.95009 0.38018
0.97874 -0.15136 -0.14814
2.24089 -0.10322 -0.23130
1.86756 0.41060 0.76682
Terms:
'x1' (column 0)
'x2' (column 1)
'x1:x2' (column 2)
상호작용을 제외한 경우에는 I()라는 연산자를 사용하여 연산과정을 명시해야 한다. 예를 들어 두 변수 x1과 x2를 더하여 새로운 데이터를 만들고 싶다면 다음과 같이 한다.
dmatrix("x1 + x2 + I(x1 + x2) + 0", df)
DesignMatrix with shape (5, 3)
x1 x2 I(x1 + x2)
1.76405 -0.97728 0.78677
0.40016 0.95009 1.35025
0.97874 -0.15136 0.82738
2.24089 -0.10322 2.13767
1.86756 0.41060 2.27816
Terms:
'x1' (column 0)
'x2' (column 1)
'I(x1 + x2)' (column 2)
선형회귀분석을 할 때는 조건수(condition number)의 영향때문에 데이터의 평균을 0으로 표준편차를 1로 만드는 스케일링(scaling) 작업을 하는 것이 분석 결과의 품질을 높일 수 있다. patsy 패키지는 스케일링을 위한 함수도 제공한다.
center(): 평균을 0으로 스케일링standardize(): 평균을 0으로하고 표준편차를 1로 스케일링scale():standardize()과 같음
예를 들어 x1 데이터의 평균을 제거하는 변환은 다음과 같다.
dm = dmatrix("center(x1) + 0", df)
dm
DesignMatrix with shape (5, 1)
center(x1)
0.31377
-1.05012
-0.47154
0.79061
0.41728
Terms:
'center(x1)' (column 0)
이 변환 연산은 다음과 같이 x1 데이터에서 x1의 평균을 빼는 것과 같다.
df.x1 - np.mean(df.x1)
0 0.313773
1 -1.050123
2 -0.471542
3 0.790613
4 0.417278
Name: x1, dtype: float64
이 때 평균값은 design_info라는 속성 내부에 저장된다.
dm.design_info.factor_infos
{EvalFactor('center(x1)'): FactorInfo(factor=EvalFactor('center(x1)'),
type='numerical',
state=<factor state>,
num_columns=1)}
이 값을 저장하는 이유는 다음과 같. 어떤 학습용 데이터를 사용하여 예측모형을 만든다고 하자. 이 때 학습성능을 좋게 하기 위해 학습용 데이터의 평균값을 데이터에서 빼는 스케일링을 실시하였다. 학습이 끝난 후 이 모형을 사용하여 실제 예측을 할 때 새로운 검증용 데이터를 이 모형에 넣으려면 모형을 학습할 때 사용한 것과 동일한 전처리를 해야 한다. 이 때 학습용 데이터의 평균값을 빼야 한다는 점에 주의한다. 이렇게 하기 위해서는 전처리 과정에서 계산한 평균값을 기억하고 있어야 한다.
sklearn.preprocessing 패키지#
sklearn패키지의 preprocessing 서브패키지도 스케일링 및 변수변환을 위한 StandardScaler 클래스는 기능을 제공한다.
이 클래스의 사용방법은 다음과 같다.
학습용 데이터를 입력으로
fit()메서드를 실행하면 평균값과 표준편차를 계산하여 객체내에 저장한다.다시 학습용 데이터를 입력으로 하여
transform()메서드를 실행하면 저장했던 평균값을 빼서 새로운 평균값이 0이 되도록 만들고, 저장한 표분편차로 나우어 새로운 표준편차가 1이 되도록 데이터를 변환하여 출력한다. 1단계와 2단계를 합쳐서fit_transform()메서드를 실행할 수도 있다.검증용 데이터를 입력으로 하여
transform()메서드를 실행해도 학습용 데이터의 평균값과 표준편차를 사용하여 검증용 데이터를 변환한다.
X = np.arange(7).reshape(-1, 1)
X
array([[0],
[1],
[2],
[3],
[4],
[5],
[6]])
from sklearn.preprocessing import StandardScaler
scaler = StandardScaler()
scaler.fit_transform(X)
array([[-1.5],
[-1. ],
[-0.5],
[ 0. ],
[ 0.5],
[ 1. ],
[ 1.5]])
그런데 만약 데이터 집합에 다른 데이터와 동떨어진 아웃라이어가 존재하는 경우에는 스케일링을 했을 때 데이터가 0이 아닌 다른 곳으로 모일 수 있다.
X2 = np.vstack([X, [[1000]]])
X2
array([[ 0],
[ 1],
[ 2],
[ 3],
[ 4],
[ 5],
[ 6],
[1000]])
scaler.fit_transform(X2)
array([[-0.38705669],
[-0.38402392],
[-0.38099115],
[-0.37795839],
[-0.37492562],
[-0.37189286],
[-0.36886009],
[ 2.64570872]])
이 때는 RobustScaler 클래스를 사용한다. 이 클래스는 중앙값이 0, IQR(interquartile range)이 1이 되도록 변환하기 때문에 아웃라이어가 섞여 있어도 대부분의 데이터는 0 주위에 남아있게 된다.
from sklearn.preprocessing import RobustScaler
scaler = RobustScaler()
scaler.fit_transform(X2)
array([[-1.00000000e+00],
[-7.14285714e-01],
[-4.28571429e-01],
[-1.42857143e-01],
[ 1.42857143e-01],
[ 4.28571429e-01],
[ 7.14285714e-01],
[ 2.84714286e+02]])
preprocessing 서브패키지는 데이터 변환을 위한 FunctionTransformer 클래스와 PolynomialFeatures 클래스도 제공한다.
PolynomialFeatures 클래스는 입력 데이터 \(x\)를 다음과 같이 여러개의 다항식으로 변환한다.
다음과 같은 입력 인수를 가진다.
degree: 차수include_bias: 상수항 생성 여부
from sklearn.preprocessing import PolynomialFeatures
poly = PolynomialFeatures(degree=2)
poly.fit_transform(X)
array([[ 1., 0., 0.],
[ 1., 1., 1.],
[ 1., 2., 4.],
[ 1., 3., 9.],
[ 1., 4., 16.],
[ 1., 5., 25.],
[ 1., 6., 36.]])
FunctionTransformer 클래스는 사용자가 지정한 함수를 사용하여 입력값 \(x\)를 변환한다.
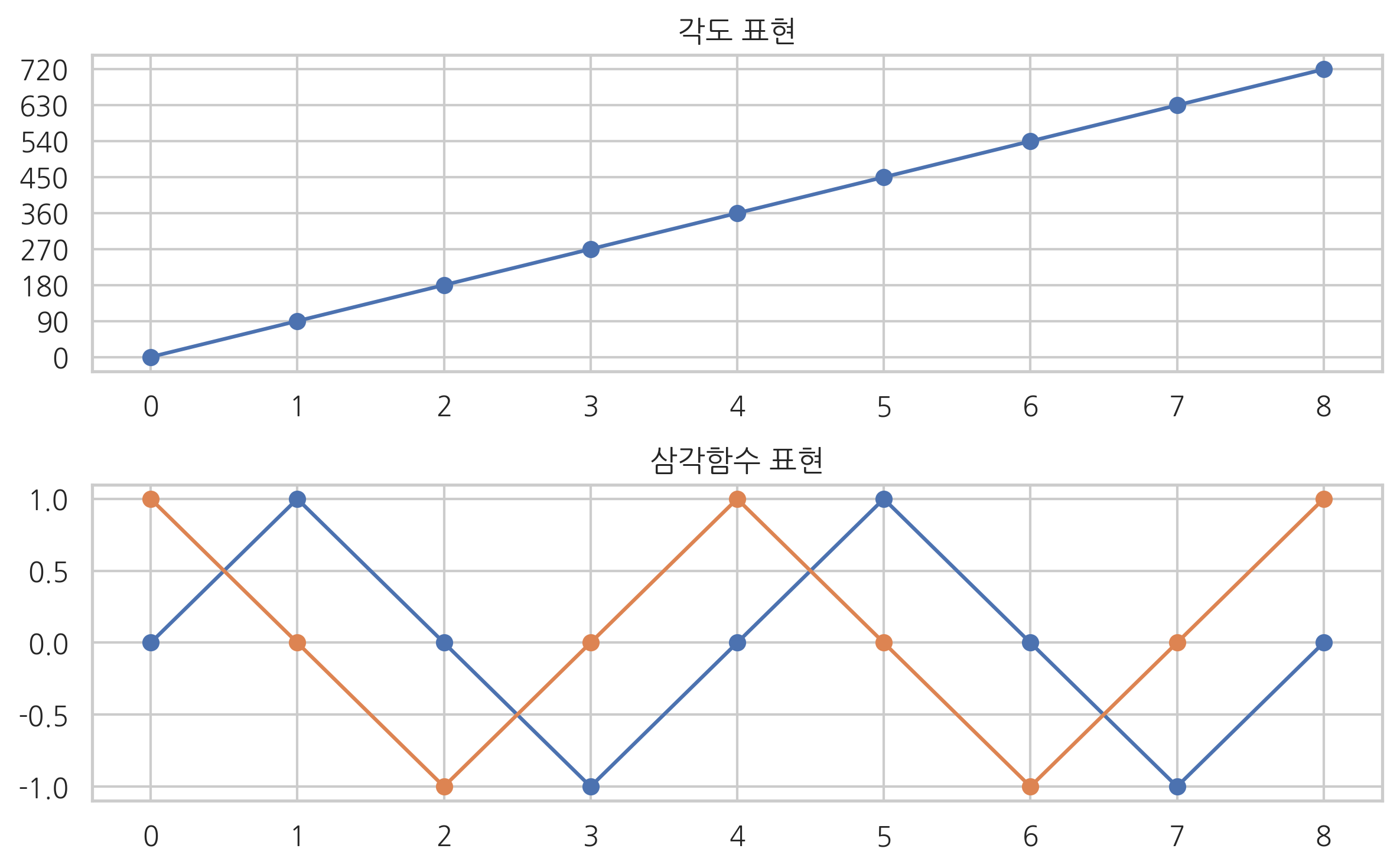
데이터 변환은 비선형 회귀분석에서 원하는 목표값을 더 잘 예측하기 위한 새로운 데이터를 만들 때 사용된다. 예를 들어 ‘360도’와 같은 각도 데이터는 그 자체로 예측문제의 입력값으로 넣을 수 없다. 0도와 360도, 10도와 370도는 사실 같은 각도지만 다른 숫자로 표현되기 때문이다. 이 때는 각도 \(\theta\)를 다음과 같이 삼각함수값의 쌍으로 바꾸면 같은 각도를 같은 숫자쌍으로 표현할 수 있다.
다음 예제 코드에서 원래 데이터 X는 각도 표시로 되어 있다.
X = 90 * np.arange(9).reshape(-1, 1)
X
array([[ 0],
[ 90],
[180],
[270],
[360],
[450],
[540],
[630],
[720]])
하지만 X2는 삼각함수로 변환되었다. 각도 0도와 각도 360도가 모두 같은 값인 (0,1)로 표현된 것을 확인할 수 있다.
from sklearn.preprocessing import FunctionTransformer
def degree2sincos(X):
x0 = np.sin(X * np.pi / 180)
x1 = np.cos(X * np.pi / 180)
X_new = np.hstack([x0, x1])
return X_new
X2 = FunctionTransformer(degree2sincos).fit_transform(X)
plt.subplot(211)
plt.plot(X, marker="o")
plt.yticks(X.flatten())
plt.title("각도 표현")
plt.subplot(212)
plt.plot(X2, marker="o")
plt.title("삼각함수 표현")
plt.tight_layout()
plt.show()