서포트 벡터 머신#
퍼셉트론은 가장 단순하고 빠른 판별 함수 기반 분류 모형이지만 판별 경계선(decision hyperplane)이 유니크하게 존재하지 않는다는 특징이 있다. 서포트 벡터 머신(SVM: support vector machine)은 퍼셉트론 기반의 모형에 가장 안정적인 판별 경계선을 찾기 위한 제한 조건을 추가한 모형이라고 볼 수 있다.

그림 45.2 : 서포트벡터머신
서포트와 마진#
다음과 같이 \(N\)개의 학습용 데이터가 있다고 하자.
판별함수모형에서 \(y\)는 \(+1\), \(-1\) 두 개의 값을 가진다
\(x\) 데이터 중에서 \(y\)값이 +1인 데이터를 \(x_+\), \(y\)값이 -1인 데이터를 \(x_-\)라고 하자.
판별함수 모형에서 직선인 판별 함수 \(f(x)\)는 다음과 같은 수식으로 나타낼 수 있다.
판별함수의 정의에 따라 \(y\) 값이 \(+1\)인 그 데이터 \(x_+\)에 대한 판별함수 값은 양수가 된다.
반대로 \(y\) 값이 \(-1\)인 그 데이터 \(x_-\)에 대한 판별함수 값은 음수가 된다.
\(y\) 값이 \(+1\)인 데이터 중에서 판별 함수의 값이 가장 작은 데이터를 \(x^+\)라고 하고 \(y\) 값이 \(-1\)인 데이터 중에서 판별함수의 값이 가장 큰 데이터를 \(x^-\)라고 하자. 이 데이터들은 각각의 클래스에 속한 데이터 중에서 가장 경계선에 가까이 붙어있는 최전방(most front)의 데이터들이다. 이러한 데이터를 서포트(support) 혹은 **서포트 벡터(support vector)**라고 한다. 물론 이 서포트에 대해서도 부호 조건은 만족되어야 한다.
서포트에 대한 판별 함수의 값 \(f(x^+)\), \(f(x^-)\) 값은 부호 조건만 지키면 어떤 값이 되어도 괜찮다. 따라서 다음과 같은 조건을 만족하도록 판별 함수를 구한다.

그림 18.6.3 : 서포트벡터의 판별함수 값
이렇게 되면 모든 \(x_+\), \(x_+\) 데이터에 대해 판별함수의 값의 절대값이 1보다 커지므로 다음 부등식이 성립한다.
판별 경계선 \(w^T x - w_0=0\) 과 점 \(x^{+}\), \(x^{-}\) 사이의 거리는 다음과 같이 계산할 수 있다.
이 거리의 합을 마진(margin)이라고 하며 마진값이 클 수록 더 경계선이 안정적이라고 볼 수 있다. 그런데 위에서 정한 스케일링에 의해 마진은 다음과 같이 정리된다.
마진 값이 최대가 되는 경우는 \(\| w \|\) 즉, \(\| w \|^2\)가 최소가 되는 경우와 같다. 즉 다음과 같은 목적함수를 최소화하면 된다.
또한 모든 표본 데이터에 대해 분류는 제대로 되어야 하므로 모든 데이터 \(x_i, y_i \; ( i = 1, \ldots, N)\)에 대해 다음 조건을 만족해야 한다. 위에서 스케일링을 사용하여 모든 데이터에 대해 \(f(x_i) = w^Tx_i - w_o\) 가 1보다 크거나 -1보다 작게 만들었다는 점을 이용한다.
라그랑주 승수법을 사용하면 최소화 목적함수를 다음과 같이 고치면 된다.
여기에서 \(a_i\)은 각각의 부등식에 대한 라그랑주 승수이다.
이 최적화 문제를 풀어 \(w\), \(w_0\), \(a\)를 구하면 판별함수를 얻을 수 있다.
KKT(Karush–Kuhn–Tucker) 조건에 따르면 부등식 제한 조건이 있는 경우에는 등식 제한조건을 가지는 라그랑주 승수 방법과 비슷하지만 \(i\)번째 부등식이 있으나 없으나 답이 같은 경우에는 라그랑지 승수의 값이 \(a_i=0\)이 된다. 이 경우는 판별함수의 값 \(w^Tx_i - w_o\)이 \(-1\)보다 작거나 \(1\)보다 큰 경우이다.
학습 데이터 중에서 최전방 데이터인 서포트 벡터가 아닌 모든 데이터들에 대해서는 이 조건이 만족되므로 서포트 벡터가 아닌 데이터는 라그랑지 승수가 0이라는 것을 알 수 있다.
듀얼 형식#
최적화 조건은 목적함수 \(L\)을 \(w\), \(w_0\)로 미분한 값이 0이 되어야 하는 것이다. $\( \dfrac{\partial L}{\partial w} = 0 \)$
이 식을 풀어서 정리하면 다음과 같아진다.
즉,
이 두 수식을 원래의 목적함수에 대입하여 \(w\), \(w_0\)을 없애면 다음과 같다.
즉,
이 때 \(a\)는 다음 조건을 만족한다.
이 문제는 \(w\)를 구하는 문제가 아니라 \(a\)만을 구하는 문제로 바뀌었으므로 듀얼형식(dual form)이라고 한다. 듀얼형식으로 바꾸면 수치적으로 박스(Box)제한 조건이 있는 이차프로그래밍(QP; quadratic programming) 문제가 되므로 원래의 문제보다는 효율적으로 풀 수 있다.
듀얼형식 문제를 풀어 함수 \(L\) 를 최소화하는 \(a\)를 구하면 예측 모형을 다음과 같이 쓸 수 있다.
\(w_0\)는
또는
또는
로 구한다.
라그랑주 승수 값이 0 즉, \(a_i = 0\) 이면 해당 데이터는 예측 모형, 즉 \(w\) 계산에 아무런 기여를 하지 않으므로 위 식을 실제로는 다음과 같다.
여기에서 \(x^T x^+\) 는 \(x\)와 \(x^+\)사이의 (코사인)유사도, \(x^T x^-\) 는 \(x\)와 \(x^-\)사이의 (코사인)유사도이므로 결국 두 서포트 벡터와의 유사도를 측정해서 값이 큰 쪽으로 판별하게 된다.
Scikit-Learn의 서포트 벡터 머신#
Scikit-Learn의 svm 서브페키지는 서포트 벡터 머신 모형인 SVC (Support Vector Classifier) 클래스를 제공한다.
from sklearn.datasets import make_blobs
X, y = make_blobs(n_samples=50, centers=2, cluster_std=0.5, random_state=4)
y = 2 * y - 1
plt.scatter(X[y == -1, 0], X[y == -1, 1], marker='o', label="-1 클래스")
plt.scatter(X[y == +1, 0], X[y == +1, 1], marker='x', label="+1 클래스")
plt.xlabel("x1")
plt.ylabel("x2")
plt.legend()
plt.title("학습용 데이터")
plt.show()
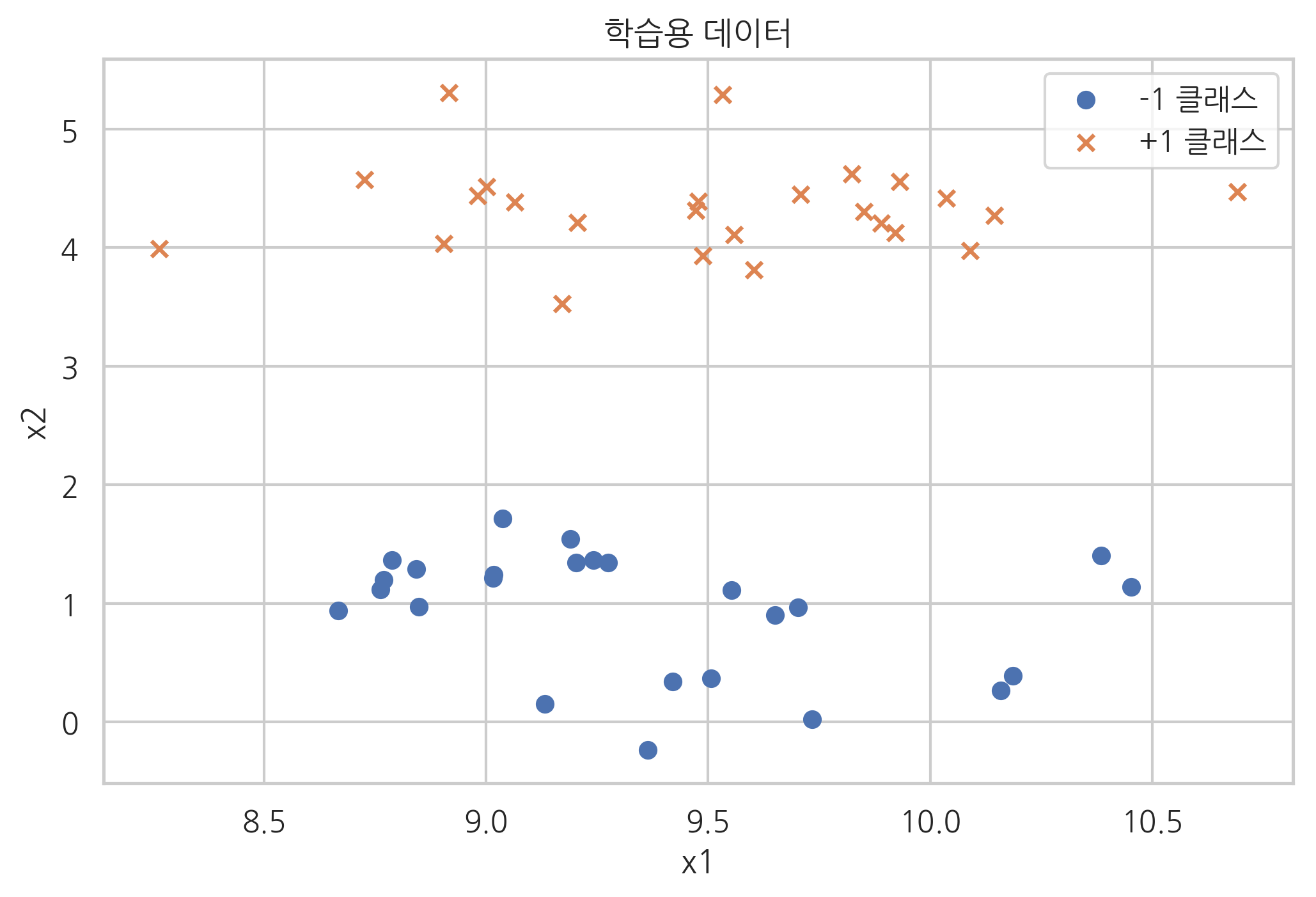
SVC 클래스는 커널(kernel)을 선택하는 인수 kernel과 슬랙변수 가중치(slack variable weight)를 선택하는 인수 C를 받는데 지금까지 공부한 서포트 벡터 머신을 사용하려면 인수를 다음처럼 넣어준다. 이 인수들에 대해서는 곧 설명한다.
SVC(kernel='linear', C=1e10)
from sklearn.svm import SVC
model = SVC(kernel='linear', C=1e10).fit(X, y)
SVC를 사용하여 모형을 구하면 다음과 같은 속성값을 가진다.
n_support_: 각 클래스의 서포트의 개수support_: 각 클래스의 서포트의 인덱스support_vectors_: 각 클래스의 서포트의 x 값. \(x^{+}\)와 \(x^{-}\)coef_: \(w\) 벡터intercept_: \(-w_0\)dual_coef_: 각 원소가 \(a_i \cdot y_i\)로 이루어진 벡터
model.n_support_
array([1, 1], dtype=int32)
model.support_
array([42, 1], dtype=int32)
model.support_vectors_
array([[9.03715314, 1.71813465],
[9.17124955, 3.52485535]])
y[model.support_]
array([-1, 1])
xmin = X[:, 0].min()
xmax = X[:, 0].max()
ymin = X[:, 1].min()
ymax = X[:, 1].max()
xx = np.linspace(xmin, xmax, 10)
yy = np.linspace(ymin, ymax, 10)
X1, X2 = np.meshgrid(xx, yy)
Z = np.empty(X1.shape)
for (i, j), val in np.ndenumerate(X1):
x1 = val
x2 = X2[i, j]
p = model.decision_function([[x1, x2]])
Z[i, j] = p[0]
levels = [-1, 0, 1]
linestyles = ['dashed', 'solid', 'dashed']
plt.scatter(X[y == -1, 0], X[y == -1, 1], marker='o', label="-1 클래스")
plt.scatter(X[y == +1, 0], X[y == +1, 1], marker='x', label="+1 클래스")
plt.contour(X1, X2, Z, levels, colors='k', linestyles=linestyles)
plt.scatter(model.support_vectors_[:, 0], model.support_vectors_[:, 1], s=300, alpha=0.3)
x_new = [10, 2]
plt.scatter(x_new[0], x_new[1], marker='^', s=100)
plt.text(x_new[0] + 0.03, x_new[1] + 0.08, "테스트 데이터")
plt.xlabel("x1")
plt.ylabel("x2")
plt.legend()
plt.title("SVM 예측 결과")
plt.show()
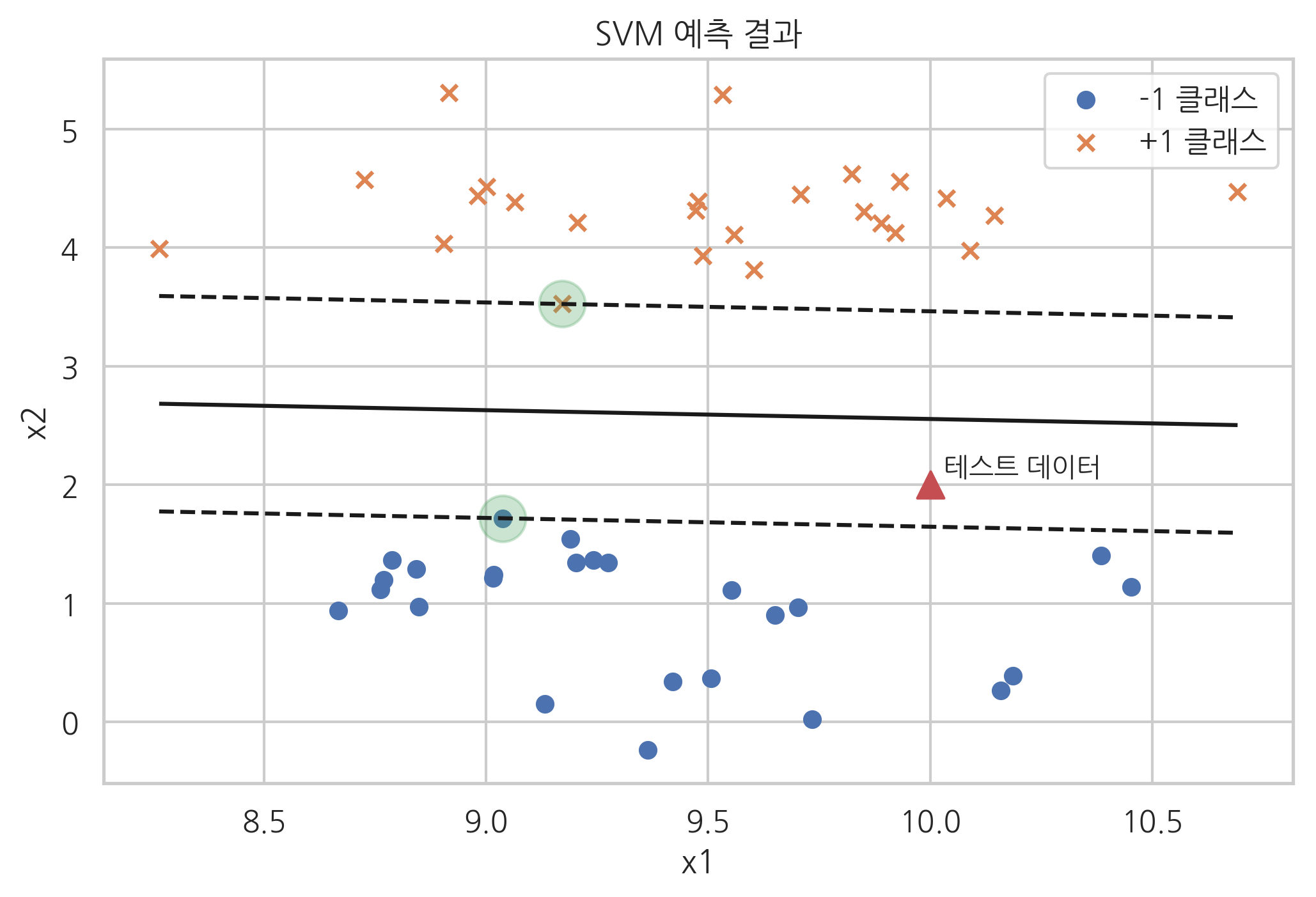
x_new = [10, 2]
model.decision_function([x_new])
array([-0.61101582])
model.coef_.dot(x_new) + model.intercept_
array([-0.61101582])
# dual_coef_ = a_i * y_i
model.dual_coef_
array([[-0.60934379, 0.60934379]])
model.dual_coef_[0][0] * model.support_vectors_[0].dot(x_new) + \
model.dual_coef_[0][1] * model.support_vectors_[1].dot(x_new) + \
model.intercept_
array([-0.61101582])
연습 문제 1#
붓꽃 문제를 서포트 벡터 머신으로 풀어보자. 다음과 같은 데이터만 사용한 이진 분류 문제로 바꾸어 풀어본다. 위의 예제와 마찬가지로 커널 인수 kernel과 슬랙변수 가중치 인수 C는 각각 linear, 1e10으로 한다.
특징 변수를 꽃받침의 길이와 폭만 사용한다.
붓꽃 종을 Setosa와 Versicolour만 대상으로 한다.
from sklearn.datasets import load_iris
iris = load_iris()
X1 = iris.data[:100, :2]
y1 = iris.target[:100]
from sklearn.svm import SVC
model1 = SVC(kernel="linear", C=1e10).fit(X1, y1)
from sklearn.metrics import classification_report
print(classification_report(y1, model1.predict(X1)))
precision recall f1-score support
0 1.00 1.00 1.00 50
1 1.00 1.00 1.00 50
accuracy 1.00 100
macro avg 1.00 1.00 1.00 100
weighted avg 1.00 1.00 1.00 100
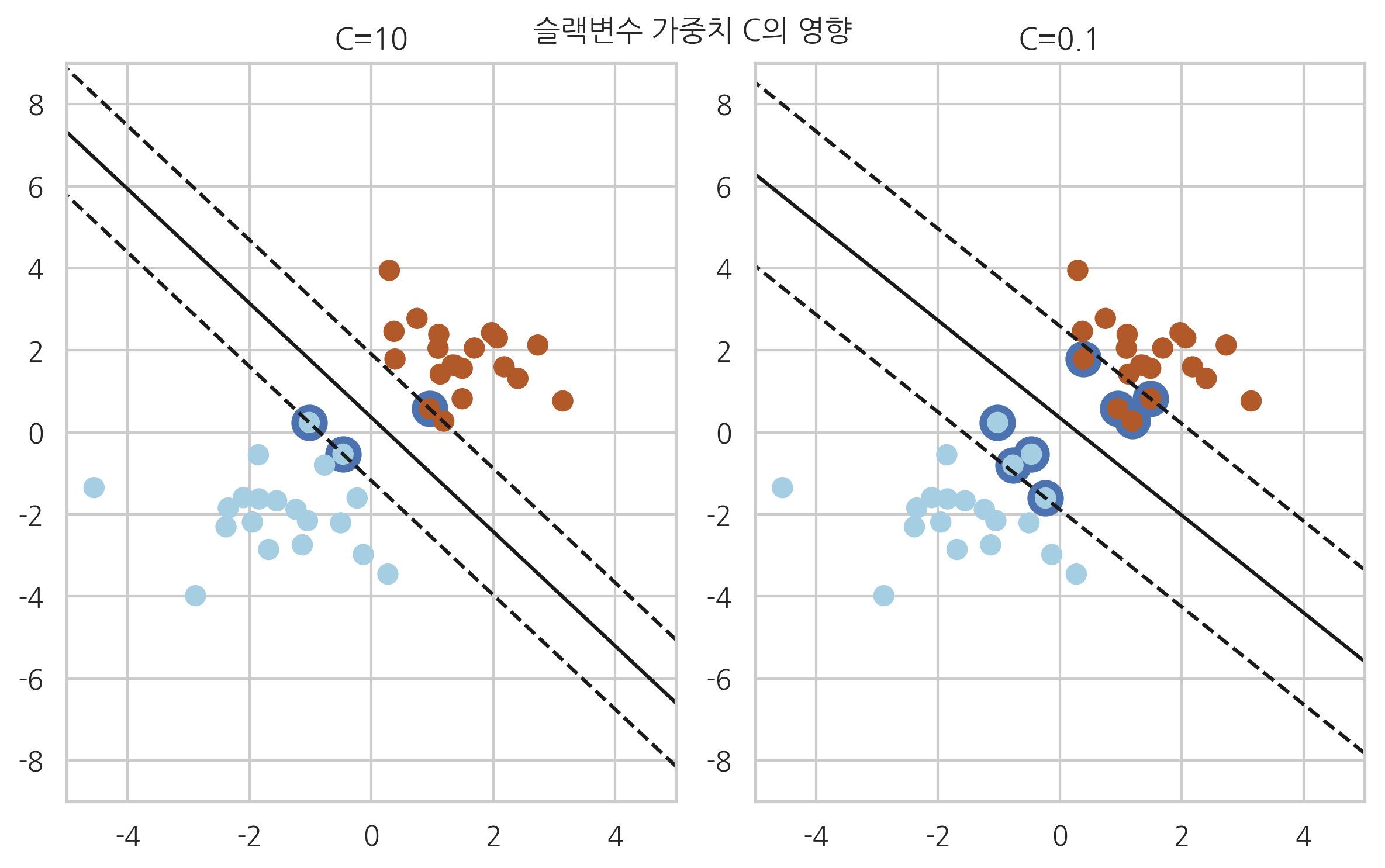
슬랙변수#
만약 데이터가 직선인 판별 경계선으로 나누어지지 않는 즉, 선형분리(linear separable)가 불가능한 경우에는 다음과 같이 슬랙변수(slack variable)를 사용하여 개별적인 오차를 허용할 수 있다.
원래 판별 함수의 값은 클래스 \(+1\) 영역의 샘플 \(x_+\)에 대해
클래스 \(-1\) 영역의 샘플 \(x_-\)에 대해
이어야 한다.
양수인 슬랙변수 \(\xi \geq 0 \)를 사용하면 이 조건을 다음과 같이 완화할 수 있다.
즉,
이 된다.
모든 슬랙변수는 0보다 같거나 크다.
위의 부등식 조건을 모두 고려한 최적화 목적함수는 다음과 같아진다.
위 식에서 \(C \sum_{i=1}^N \xi_i\) 항은 슬랙변수의 합이 너무 커지지 않도록 제한하는 역할을 한다.

그림 18.6.3 : \(C\) 값에 따른 판별 함수의 변화
np.random.seed(0)
X = np.r_[np.random.randn(20, 2) - [2, 2], np.random.randn(20, 2) + [2, 2]]
Y = [-1] * 20 + [1] * 20
plotnum = 1
for name, penalty in (('C=10', 10), ('C=0.1', 0.1)):
clf = SVC(kernel='linear', C=penalty).fit(X, Y)
xx = np.linspace(-5, 5)
x_jin = -5
x_jax = 5
y_jin = -9
y_jax = 9
XX, YY = np.mgrid[x_jin:x_jax:200j, y_jin:y_jax:200j]
levels = [-1, 0, 1]
linestyles = ['dashed', 'solid', 'dashed']
Z = clf.decision_function(np.c_[XX.ravel(), YY.ravel()])
Z = Z.reshape(XX.shape)
plt.subplot(1, 2, plotnum)
plt.contour(XX, YY, Z, levels, colors='k', linestyles=linestyles)
plt.scatter(clf.support_vectors_[:, 0], clf.support_vectors_[:, 1], s=120, linewidth=4)
plt.scatter(X[:, 0], X[:, 1], c=Y, s=60, linewidth=1, cmap=plt.cm.Paired)
plt.xlim(x_jin, x_jax)
plt.ylim(y_jin, y_jax)
plt.title(name)
plotnum += 1
plt.suptitle("슬랙변수 가중치 C의 영향")
plt.tight_layout()
plt.show()
연습 문제 2#
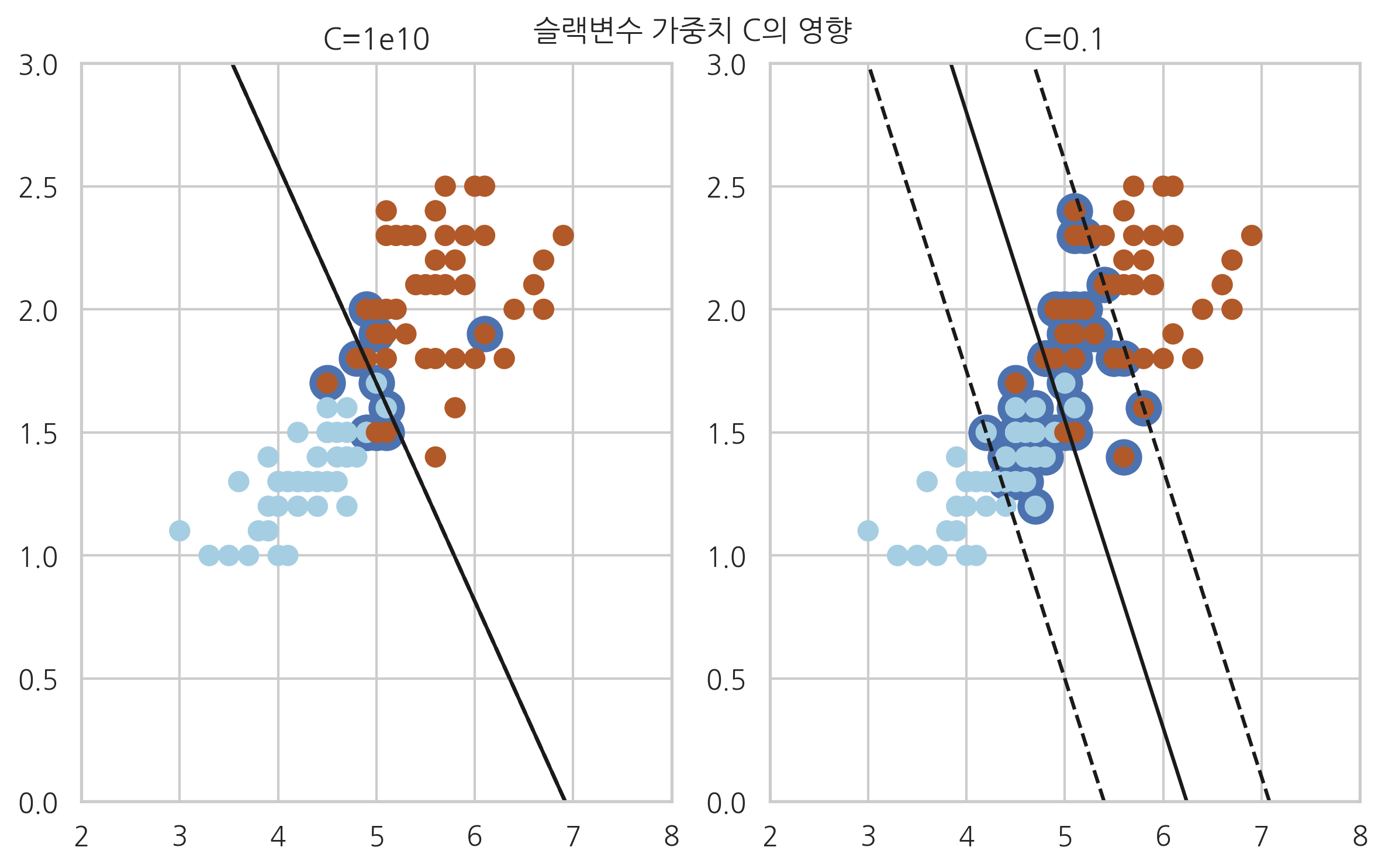
붓꽃 문제를 서포트 벡터 머신으로 풀어보자. 다음과 같은 데이터만 사용한 이진 분류 문제로 바꾸어 풀어본다. 위의 예제와 마찬가지로 커널 인수 kernel는 각각 linear로 한다. 슬랙변수 가중치 인수 C를 여러가지 값으로 바꾸어 보면서 서포트가 어떻게 바뀌는지 살펴본다.
특징 변수를 꽃잎의 길이와 폭만 사용한다.
붓꽃 종을 Virginica와 Versicolour만 대상으로 한다.
from sklearn.datasets import load_iris
iris = load_iris()
X2 = iris.data[50:, 2:]
y2 = iris.target[50:] - 1
from sklearn.svm import SVC
model1 = SVC(kernel="linear", C=1e10).fit(X2, y2)
from sklearn.metrics import classification_report
print(classification_report(y2, model1.predict(X2)))
precision recall f1-score support
0 0.91 1.00 0.95 50
1 1.00 0.90 0.95 50
accuracy 0.95 100
macro avg 0.95 0.95 0.95 100
weighted avg 0.95 0.95 0.95 100
plotnum = 1
for name, penalty in (('C=1e10', 1e10), ('C=0.1', 0.1)):
clf = SVC(kernel='linear', C=penalty).fit(X2, y2)
xx = np.linspace(2, 8)
x_jin = 2
x_jax = 8
y_jin = 0
y_jax = 3
XX, YY = np.mgrid[x_jin:x_jax:200j, y_jin:y_jax:200j]
levels = [-1, 0, 1]
linestyles = ['dashed', 'solid', 'dashed']
Z = clf.decision_function(np.c_[XX.ravel(), YY.ravel()])
Z = Z.reshape(XX.shape)
plt.subplot(1, 2, plotnum)
plt.contour(XX, YY, Z, levels, colors='k', linestyles=linestyles)
plt.scatter(clf.support_vectors_[:, 0], clf.support_vectors_[:, 1], s=120, linewidth=4)
plt.scatter(X2[:, 0], X2[:, 1], c=y2, s=60, linewidth=1, cmap=plt.cm.Paired)
plt.xlim(x_jin, x_jax)
plt.ylim(y_jin, y_jax)
plt.title(name)
plotnum += 1
plt.suptitle("슬랙변수 가중치 C의 영향")
plt.tight_layout()
plt.show()
연습 문제 3#
MNIST Digit Image 분류 문제를 서포트 벡터 머신으로 풀어보자.
얼굴 이미지 인식#
from sklearn.datasets import fetch_olivetti_faces
faces = fetch_olivetti_faces()
N = 2
M = 5
np.random.seed(0)
fig = plt.figure(figsize=(9, 5))
plt.subplots_adjust(top=1, bottom=0, hspace=0, wspace=0.05)
klist = np.random.choice(range(len(faces.data)), N * M)
for i in range(N):
for j in range(M):
k = klist[i * M + j]
ax = fig.add_subplot(N, M, i * M + j + 1)
ax.imshow(faces.images[k], cmap=plt.cm.bone)
ax.grid(False)
ax.xaxis.set_ticks([])
ax.yaxis.set_ticks([])
plt.title(faces.target[k])
plt.tight_layout()
plt.show()

from sklearn.model_selection import train_test_split
X_train, X_test, y_train, y_test = train_test_split(faces.data, faces.target, test_size=0.4, random_state=0)
from sklearn.svm import SVC
svc = SVC(kernel='linear').fit(X_train, y_train)
N = 2
M = 5
np.random.seed(4)
fig = plt.figure(figsize=(9, 5))
plt.subplots_adjust(top=1, bottom=0, hspace=0, wspace=0.05)
klist = np.random.choice(range(len(y_test)), N * M)
for i in range(N):
for j in range(M):
k = klist[i * M + j]
ax = fig.add_subplot(N, M, i * M + j + 1)
ax.imshow(X_test[k:(k + 1), :].reshape(64, 64), cmap=plt.cm.bone)
ax.grid(False)
ax.xaxis.set_ticks([])
ax.yaxis.set_ticks([])
plt.title("%d => %d" %
(y_test[k], svc.predict(X_test[k:(k + 1), :])[0]))
plt.tight_layout()
plt.show()

from sklearn.metrics import classification_report, accuracy_score
y_pred_train = svc.predict(X_train)
y_pred_test = svc.predict(X_test)
print(classification_report(y_train, y_pred_train))
precision recall f1-score support
0 1.00 1.00 1.00 4
1 1.00 1.00 1.00 5
2 1.00 1.00 1.00 6
3 1.00 1.00 1.00 8
4 1.00 1.00 1.00 8
5 1.00 1.00 1.00 5
6 1.00 1.00 1.00 4
7 1.00 1.00 1.00 7
8 1.00 1.00 1.00 8
9 1.00 1.00 1.00 7
10 1.00 1.00 1.00 4
11 1.00 1.00 1.00 6
12 1.00 1.00 1.00 6
13 1.00 1.00 1.00 6
14 1.00 1.00 1.00 4
15 1.00 1.00 1.00 4
16 1.00 1.00 1.00 8
17 1.00 1.00 1.00 4
18 1.00 1.00 1.00 9
19 1.00 1.00 1.00 4
20 1.00 1.00 1.00 9
21 1.00 1.00 1.00 6
22 1.00 1.00 1.00 7
23 1.00 1.00 1.00 5
24 1.00 1.00 1.00 6
25 1.00 1.00 1.00 5
26 1.00 1.00 1.00 5
27 1.00 1.00 1.00 8
28 1.00 1.00 1.00 6
29 1.00 1.00 1.00 4
30 1.00 1.00 1.00 6
31 1.00 1.00 1.00 5
32 1.00 1.00 1.00 6
33 1.00 1.00 1.00 7
34 1.00 1.00 1.00 4
35 1.00 1.00 1.00 7
36 1.00 1.00 1.00 6
37 1.00 1.00 1.00 6
38 1.00 1.00 1.00 9
39 1.00 1.00 1.00 6
micro avg 1.00 1.00 1.00 240
macro avg 1.00 1.00 1.00 240
weighted avg 1.00 1.00 1.00 240
print(classification_report(y_test, y_pred_test))
precision recall f1-score support
0 0.86 1.00 0.92 6
1 1.00 1.00 1.00 5
2 1.00 1.00 1.00 4
3 0.50 1.00 0.67 2
4 1.00 0.50 0.67 2
5 1.00 1.00 1.00 5
6 0.83 0.83 0.83 6
7 1.00 0.67 0.80 3
8 0.67 1.00 0.80 2
9 1.00 1.00 1.00 3
10 1.00 1.00 1.00 6
11 1.00 1.00 1.00 4
12 0.67 1.00 0.80 4
13 1.00 1.00 1.00 4
14 1.00 1.00 1.00 6
15 1.00 0.33 0.50 6
16 0.67 1.00 0.80 2
17 1.00 1.00 1.00 6
18 1.00 1.00 1.00 1
19 1.00 1.00 1.00 6
20 1.00 1.00 1.00 1
21 1.00 0.75 0.86 4
22 1.00 1.00 1.00 3
23 0.71 1.00 0.83 5
24 1.00 1.00 1.00 4
25 1.00 1.00 1.00 5
26 1.00 1.00 1.00 5
27 1.00 1.00 1.00 2
28 1.00 1.00 1.00 4
29 1.00 1.00 1.00 6
30 1.00 1.00 1.00 4
31 1.00 1.00 1.00 5
32 1.00 1.00 1.00 4
33 1.00 1.00 1.00 3
34 1.00 0.83 0.91 6
35 1.00 0.67 0.80 3
36 1.00 1.00 1.00 4
37 1.00 1.00 1.00 4
38 0.50 1.00 0.67 1
39 0.67 0.50 0.57 4
micro avg 0.93 0.93 0.93 160
macro avg 0.93 0.93 0.91 160
weighted avg 0.95 0.93 0.92 160